Risale agli albori delle tecniche di biologia molecolare il tentativo di ottenere risultati da singole cellule. I primi autori, analizzarono 226 cellule buccali da differenti individui e isolando ciascuna cellula con procedure di micromanipolazione. Ottennero circa il 50% di profili genetici completi e un elevato numero di profili parziali, comunque interpretabili [1]. Questi studi spalancarono le porte alla possibilità di analizzare tracce veramente ridotte di materiale genetico. Per usare le parole degli autori, «impronte digitali imbrattate, singole scaglie di forfora, spermi singoli in casi di violenze multiple ed esigui campioni lasciati su armi o veicoli».
Effettivamente, profili del DNA possono essere ottenuti da oggetti che siano stati toccati anche per breve tempo. Quando le cellule sono poche e i nuclei esigui, si parla di analisi con basso numero di copie (low-level DNA, low copy number LCN, low-template LT). Presentano un alto livello di difficoltà, producendo talvolta risultati controversi. In senso più generale il termine trace-DNA, DNA-traccia, definisce tutti quei campioni che rientrano al di sotto delle soglie raccomandate per ogni fase analitica [2].
In Gran Bretagna per esempio ci fu l’interruzione delle analisi LCN dopo l’attentato di Omagh nel 1998. Il Forensic Science Service britannico aveva introdotto le tecniche per queste analisi applicandole fino a quel momento in oltre 21.000 casi. Rifiutò tuttavia di rivelarne nel dettaglio le procedure analitiche adottate [3]. Un comitato di esperti per conto del Ministero degli Interni britannico ritenne la metodica robusta e adatta allo scopo e dunque fu reintrodotta dal 2009. L’FBI nel 2001 ha raccomandato di farne uso solo in casi particolari, per esempio per l’identificazione di resti scheletrici di persone scomparse [4]. Negli Stati Uniti sono state pronunciate numerose sentenze discordanti riguardo all’ammissibilità in processo di analisi basate sul basso numero di copie. Manca un accordo nella comunità scientifica riguardo a procedure e interpretazione statistica.
Esiguità delle tracce, presenza di contaminanti e inibitori, degradazione ambientale sono tutte cause per le quali gli esperimenti possono produrre profili genetici parziali o incompleti. Nei quali non è univoca l’interpretazione del corretto profilo genetico e per i quali, talvolta, è impossibile distinguere gli artefatti dal DNA originale [5].
In alcuni casi non si ottiene informazione da un certo locus. In altri si possono verificare fenomeni di assenza di forme alleliche (drop-out), oppure la loro comparsa (drop-in), comunque assenti nel campione originario.
Vari esami sperimentali dimostrano che tali campioni, pur derivando da una medesima origine, possono dare risultati analitici diversi. Il che è a dir poco inquietante parlando di identificazione personale.
Il motivo di questi risultati è imputabile alla mancanza di accuratezza del metodo di amplificazione enzimatica in vitro. Avviene a produzione di prodotti di amplificazione che non riproducono esattamente il genotipo del campione originario. Del resto, sono fenomeni che avvengono anche nel nostro corpo, durante la riproduzione cellulare. Ma il nostro organismo è in grado di attuare diversi metodi di riparazione riguardo a tali difetti replicativi. Vari meccanismi mettono in azione complessi enzimatici specifici riparativi.
Lo stesso non avviene nel metodo artificiale sostanzialmente semplice, contenendo lo stretto necessario per la duplicazione del DNA. Sperimentalmente si è però osservato che gli errori dell’enzima DNA polimerasisono aspecifici, quindi casuali, e si manifestano con una probabilità non superiore allo 0,3%. In altri termini, se una forma allelica è davvero presente in un estratto ottenuto da una traccia, è prevedibile osservarla se l’esperimento sarà ripetuto. Viceversa, se un allele si è generato per un errore, sarà improbabile visualizzarlo in due esperimenti indipendenti.
Su questo assunto si sono sviluppate tecniche analitiche specifiche, condivise anche con l’emissione di linee guida da parte della comunità forense [6]. Ad esempio, il metodo suggerito da Caragine et al. [7] è basato sulla ripetizione dell’amplificazione di un campione trace-DNA, determinando un cosiddetto «profilo consenso». Ciascuna forma allelica viene registrata nel profilo finale se osservata almeno due volte in amplificazioni ripetute. Un eventuale omozigote viene indicato con una sigla «Z» per evidenziare l’eventualità che il campione fosse in realtà eterozigote. In questo caso si suppone che un allele non sia amplificato per difetto della PCR.
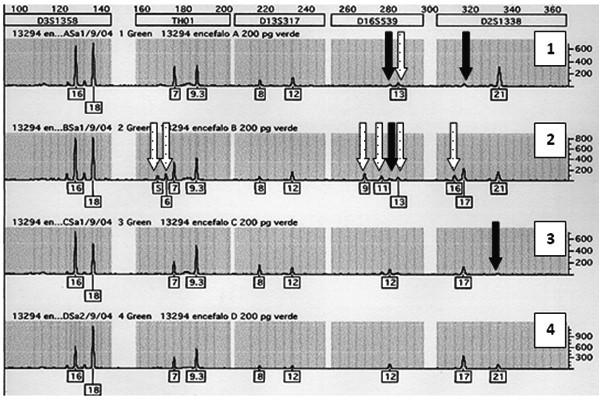
Gli elettroferogrammi sono l’esito di una sperimentazione. Un frammento di tessuto integro (profilo 4) e tre frammenti dello stesso campione degradati artificialmente con calore (1), umidità (2), ioni metallici (3). Le frecce bianche indicano alleli aggiuntivi (drop-in), quelle nere gli alleli mancanti (drop-out), rispetto al campione non degradato. Da: U. Ricci, Limiti e aspettative della genetica forense, in C. Conti (ed.), Scienza e processo penale. Nuove frontiere e vecchi pregiudizi, Giuffrè, Milano 2011, 247-262.
I profili generati devono essere interpretati dopo che si è verificata la possibilità di replicare un risultato in amplificazioni successive e indipendenti.
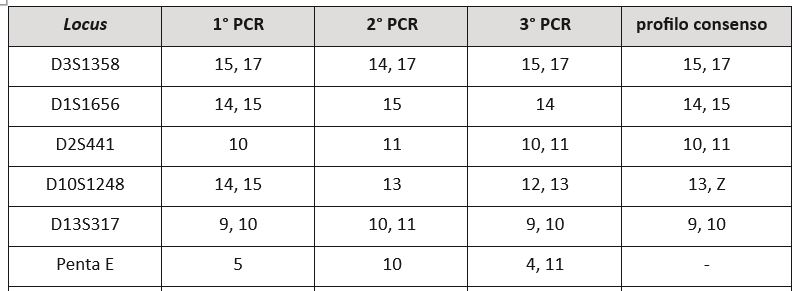
La tabella mostra il risultato analitico dell’esame di una traccia con basso numero di copie di DNA. Tre amplificazioni indipendenti generano un profilo consenso. Quest’ultimo può essere utilizzato per le comparazioni con profili di soggetti sospettati di aver lasciato la traccia.
È evidente che un laboratorio che decida di effettuare questi esami deve adottare particolari cautele rispetto ai campioni ordinari. A questo proposito, i sistemi di validazione del metodo in accordo con la ISO/IEC 17025 sono essenziali. Particolare cura deve essere poi riposta al controllo delle contaminazioni. Per esempio con la scrupolosa rimozione della contaminazione intra-laboratorio, possibilmente utilizzando aria filtrata, frequenti cambi dei guanti e decontaminazioni accurate dei materiali di laboratorio.
Generare profili a partire da poche copie di DNA pone poi il problema di come interpretare i profili genetici risultanti. Peter Gill, per esempio ha proposto una definizione alternativa di trace-DNA in un modo che lui stesso definisce, volutamente, vago. «È definito trace-DNA ogni campione nel quale vi sia incertezza riguardo al fatto che possa essere associato con lo stesso evento criminale, così che sia possibile che il trasferimento possa essere avvenuto prima del delitto (innocent transfer) o dopo il delitto (investigator mediated)».
La materia in Italia è stata associata, tra i casi pratici più eclatanti, all’omicidio di Meredith Kercher a Perugia. Due reperti fondamentali per sostenere la tesi accusatoria furono un coltello e un gancetto di un reggiseno. Su questi furono identificati profili genetici dovuti a trace-DNA. Oltre all’interesse mediatico, l’episodio fu un’occasione di confronto per gli esperti della materia. Con una vera e propria mobilitazione dei più eminenti scienziati forensi di tutto il mondo. In un convegno internazionale tenutosi a Roma [8], furono discussi i vari aspetti legati alla materia dell’analisi trace-DNA.
La determinazione di profili del DNA da questi campioni, se effettuati secondo le regole stabilite dal laboratorio in accordo al consenso internazionale, produce effettivamente risultati robusti la cui utilità nei casi reali è talvolta tangibile, con riscontri positivi dal punto di vista identificativo.
[1] Cf. R.A.H. van Oorschot – M.J. Jones, DNA fingerprinting from fingerprints, in Nature (1997)387, 767; I. Findlay et al., DNA fingerprinting from single cells, in Nature (1997)389, 555-556.
[2] R.A.H. van Oorschot – K.N. Ballantyne – R.J. Mitchell, Forensic trace DNA: a review, in Investig Genet (2010)1, 14.
[3] P. Gill – J. Buckleton, A universal strategy to interpret DNA profiles that does not require a definition of low-copy-number, in Forensic Sci Int Genet (2010), 221-227.
[4] B. Budowle et al., Validity of Low Copy Number Typing and Applications to Forensic Science, in Croat Med J (2009)50, 207-217.
[5] P. Fattorini et al., Estimating the integrity of aged DNA samples by CE, in Electrophoresis (2009)30, 3986-3995.
[6] P. Gill et al., DNA commission of the International Society of Forensic Genetics: Recommendations on the evaluation of STR typing results that may include drop-out and/or drop-in using probabilistic methods, in Forensic Sci Int: Genetics (2012)6, 679-688.
[7] T. Caragine et al., Validation of testing and interpretation protocols for low template DNA samples using AmpFlSTR Identifiler, in Croat Med J (2009)50/3, 250-267.
[8] V. Pascali – M. Prinz, Highlights of the conference ‘The hidden side of DNA profiles: Artifacts, errors and uncertain evidence’, in Forensic Sci Int: Genetics (2012)6, 775–777.
