All’interno del citoplasma cellulare sono presenti molti mitocondri, le cosiddette «centrali energetiche» della cellula, perché al loro interno si realizzano fondamentali trasformazioni biochimiche che permettono di immagazzinare energia, sotto forma di molecole di adenosina trifosfato (ATP). Ciascun mitocondrio costituisce un’unità autonoma e per questo possiede al proprio interno più molecole, fino a un centinaio, di un proprio, esclusivo, materiale genetico: il DNA mitocondriale (mtDNA).
Il mtDNA è molto più semplice di quello presente nel nucleo e molto più corto, costituito soltanto da 16.569 paia di basi a formare una doppia elica circolare, contenente pochi geni.
Per l’identificazione umana viene studiata una zona detta d-loop, comprendente le regioni ipervariabili HV1 e HV2, soggette ad ampia variabilità nella popolazione e al cui interno sono presenti molti polimorfismi di sequenza [1]. La regione copre circa 1100 bp in una zona non codificante del genoma.
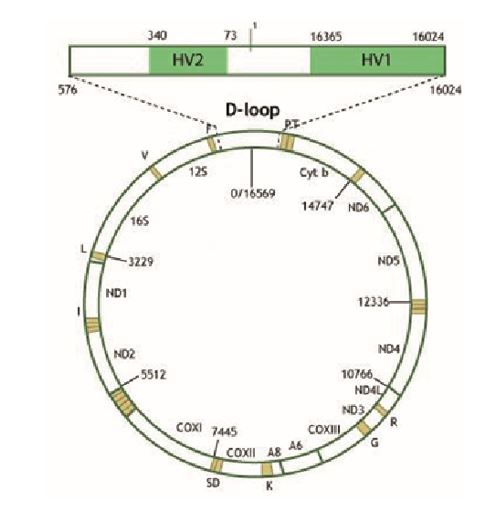
L’esistenza del DNA mitocondriale supporta l’affascinante teoria dell’endosimbiosi, secondo la quale le cellule degli organismi superiori apparvero per la prima volta quando una cellula batterica venne assorbita da un’altra cellula, senza essere digerita. In questo modo si sarebbe formato il primo organismo pluricellulare. Questa teoria è supportata da numerose osservazioni tra le quali la notevole similitudine dell’mtDNA con il DNA batterico, l’assenza di istoni e una capacità di replicazione aumentata e autonoma rispetto alla cellula ospite. Il mitocondrio è comunque fondamentale per la cellula, contribuendo con 13 mRNA, 22 tRNA e 2 rRNA.
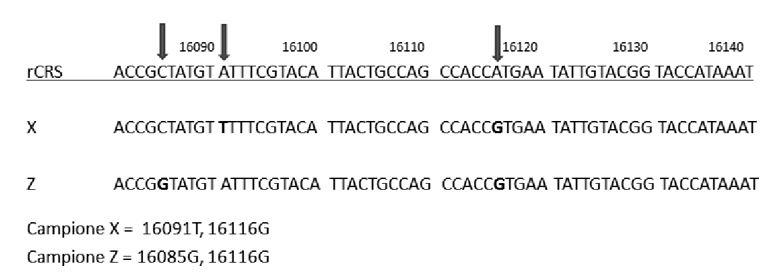
Le sequenze mitocondriali vengono classificate in base alle differenze con una sequenza di riferimento (rCRS) [2] e anche questo rappresenta una sorta di semplificazione, perché invece di trascrivere più di 1.000 lettere è sufficiente registrare le variazioni osservate, come illustrato a fianco.
In questo caso, quindi, la differenza tra individui si coglie da variazioni di singole basi tra regioni analoghe.
La disponibilità delle recenti metodiche di sequenziamento massivo permette tuttavia, in tempi e con costi ragionevoli, la determinazione dell’intera sequenza mitocondriale, un approccio che ovviamente permette di individuare una variabilità ancora maggiore rispetto a quella della sola regione del d-loop.
Una caratteristica dell’mtDNA è quella di essere ereditato per via materna, perché gli spermatozoi non contengono praticamente mitocondri. Questo peculiare meccanismo di trasmissione viene molto utilizzato per studi di tipo filogenetico, perché mentre all’eredità nucleare hanno contribuito più soggetti (i genitori, i nonni, i bisnonni, i trisavoli, ecc), la linea di discendenza mitocondriale è dovuta a una sola antenata che ha trasmesso il proprio mtDNA praticamente inalterato.
Per questo è comune dire che possediamo l’mtDNA dell’Eva primordiale.
L’analisi dell’mtDNA è utile perché:
- ha una sensibilità molto superiore rispetto alla determinazione di profili genetici nucleari, consentendo quindi di analizzare anche campioni molto danneggiati o con poche cellule, come per esempio capelli privi di bulbo;
- può essere impiegata per effettuare test di maternità, visto il tipo particolare di trasmissione;
consente di fare inferenze sull’origine geografica del donatore di un campione, perché ciascun profilo mitocondriale corrisponde a un particolare aplogruppo (raggruppamenti di mutazioni genetiche), che può essere quindi ricondotto a un progenitore ancestrale.
Per questo è comune dire che possediamo l’mtDNA dell’Eva primordiale.
A differenza di quanto avviene per il materiale genetico nucleare, è possibile che in una stessa persona coesistano diverse popolazioni di DNA mitocondriale, evidenza descritta in campioni di sangue e nelle formazioni pilifere [3][1].
L’analisi dell’mtDNA richiede comunque estrema attenzione e cautele maggiori di quelle attuate per le analisi dei campioni ordinari, vista l’altissima probabilità di introdurre contaminazioni nella fase analitica [4][2].
Il mitocondrio è poi ricco di altre informazioni.
L’analisi di specie
Sulla sua superficie sono presenti due geni molto importanti, il citocromo B (Cyt B) e il citocromo C (COI), entrambi implicati nella catena di trasporto degli elettroni. Si tratta di geni molto conservati tra le specie, nel senso che nel corso delle generazioni non hanno subìto particolari modifiche (note con il termine di mutazioni). Questo significa che ogni organismo vivente ha avuto cura di tenere ben conservati nella propria «scatola degli attrezzi genetica» questi due geni, non modificandoli, perché estremamente utili per la vita della cellula.
In natura questa è una regola ferrea: non vi è alcun motivo di modificare ciò che funziona bene [5][3]! Quindi, fin dall’epoca primordiale, ogni organismo ha conservato, praticamente inalterati, alcuni geni con caratteristiche specifiche, ancora più evidenti qualora si confrontino specie molto affini. Per esempio, le differenze nel citocromo tra l’uomo e la scimmia riguardano solo il sito 66 (isoleucina nel COI dell’uomo e treonina nella scimmia). Si tratta quindi che si tratta di una proteina a evoluzione lenta.
Sia il gene Cyt B che il gene COI sono utili perché:
- contengono sufficiente variabilità tra gruppi animali;
- sono geni corti, tali da poter essere sequenziati in una sola reazione;
- contengono regioni conservate di complementazione con primer universali.
Le raccomandazioni dell’ISFG [6] indicano che la scelta preferenziale è per questi due marcatori, anche se il COI è forse l’esempio più illustrativo. Ciò grazie ai lavori pionieristici di Fitch e Margoliash [7]. Infatti è utilizzato per la tassonomia degli organismi viventi, attraverso la cosiddetta analisi DNA barcode [8].
Per la ricerca di similarità di sequenze sono disponibili numerose risorse sul web. Una delle più note è costituita dall’Universal Protein Resource (Banca dati disponibile al sito http://www.uniprot.org/).
Le applicazioni sono le più varie. Dall’individuazione delle frodi alimentari alla caratterizzazione di specie in reperti forensi, a quella dei patogeni per l’uomo. In particolare di quelli utilizzati per il bioterrorismo, come l’antrace e il botulino [9].
Nel giugno 2013 l’autorità giudiziaria dispose un accertamento su alcune partite di pesce congelato. Conservate in una cella frigorifera di un ingrosso si voleva capire se corrispondessero effettivamente a quanto dichiarato sui confezionamenti. Si trattava di diversi quintali di pescato. Fu necessaria la presenza di personale della ASL per procedere a un campionamento secondo le direttive di legge.

L’esame fu effettuato sequenziando le regioni dei citocromi B e C, dimostrando che nessuna delle specie era quella dichiarata dal venditore. La partita di pesce sciabola era invece pesce coltello (Trichiurus lepturus). Quella di anguilla seccata risultò murena (Muraenesox bagio) e quella di corvina era invece ombrina giapponese (Miichthys miiuy). Bello sapere cosa mangiamo, vero?
[1] S. Anderson et al., Sequence and organization of the human mitochondrial genome, in Nature (1981)90, 457-465.
[2] R.M. Andrews et al., Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA, in Nat Genet (1999)23, 147.
[3] G.G. Paneto et al., Heteroplasmy in hair: study of mitochondrial DNA third hypervariable region in hair and blood samples, J Forensic Sci (2010)55/3, 715-718.
[4] W. Parson et al., DNA Commission of the International Society for Forensic Genetics: revised and extended guidelines for mitochondrial DNA typing, in Forensic Sci Int: Genetics (2014)13, 134-142.
[5] R. Dawkins, Il Gene egoista, Mondadori, Milano 2014. L’argomento è in verità complesso ed è ancora oggetto di dibattiti e prese di posizione diverse. È il gene che si perpetua per se stesso o è l’organismo a mantenerlo perché utile alla vita dell’intero organismo? Il libro è uscito nel 1976 e dopo quello ne sono usciti altri. Dice l’autore: «L’argomento base di questo libro è che noi, e tutti gli altri animali, siamo macchine create dai nostri geni» per la loro sopravvivenza e proliferazione e che «una qualità predominante da aspettarsi in un gene che abbia successo è un egoismo spietato». Ovviamente questo non significa che l’egoismo del gene si trasmetta pari pari all’individuo né che ci sia tra loro un rapporto causale diretto. Anzi, spesso l’egoismo del gene si manifesta a livello di individui o di gruppi con forme di comportamento che possiamo tranquillamente definire altruistiche.
[6] A. Linacre et al., ISFG: recommendation regarding the use of non-human (animal) DNA in forensic genetic investigations, in Forensic Sci Int: Genetics (2011)5, 501-505.
[7] E. Margoliash – W.M. Fitch, Evolutionary variability of cytochrome C primary structures, in Annals of the New York Academy of Sciences (1968)51, 359-381.
[8] P.D. Hebert et al., Biological identification through DNA barcode, in Proc Biol Sci (2003)270, 313-321.
[9] U. Ricci, Descrizione di un metodo per l’identificazione di specie in genetica forense, in Rivista Italiana di Medicina Legale (2013)2, 718-731.
