Determinata la natura del reperto biologico, quindi identificata la traccia, si vuole adesso individualizzarla, cioè rilevare quelle caratteristiche di diversità che la rendono unica.
La determinazione di profili genetici da campioni e reperti è proprio il tema centrale della legge sulla Banca dati che tocca nella disciplina attuativa anche il tema operativo, in tutto il capo IV, «Tecniche, modalità di analisi e conservazione dei campioni biologici e tempi di conservazione dei campioni biologici e dei profili del DNA».
Quanto finora si è detto sul reperto (fotografie, prelievi, esami speditivi, ecc.) è dunque propedeutico agli esami veri e propri sul DNA. La comprensione delle attività preliminari finora descritte accede a settori della conoscenza relativamente comuni per ognuno di noi. Chi non ha mai provato, magari durante il periodo scolastico, a guardare qualche capello o insetto al microscopio, a sottoporsi a un prelievo oppure a guardare una lastra radiografica? In tutti questi casi si ha una percezione diretta della natura del campione biologico oppure, seppur vaga, anche di quello che può essere un risultato di laboratorio quando si tenti di interpretare il risultato degli esami del sangue o si eseguano, non senza una certa trepidazione, valutazioni sulla positività o meno di una lastrina per un test di gravidanza.
Riguardo alla biologia molecolare, queste conoscenze non sono più sufficienti. Volendo studiare il DNA, entità così infinitesimale, si deve compiere uno sforzo che è quasi di «fede» per capire che cosa si fa, utilizzando tecnologie altamente sofisticate tipiche di un laboratorio di genetica molecolare.
Mi è però capitato di tenere un paio di lezioni, in parte sperimentali, a studenti delle scuole medie superiori e, al momento di prepararle, ho trovato su internet una ricchezza di informazioni che non conoscevo e nelle quali, tra l’altro, si mostrava come estrarre DNA dalla frutta[1]. È probabile che dare un’occhiata a questi siti agevoli il lettore nella comprensione delle varie procedure che saranno di seguito descritte e che porteranno a ottenere il materiale genetico necessario per lo studio dei polimorfismi genetici.

Nell’immagine a fianco sono rappresentate numerose cellule epiteliali. Noterete dei puntini che sono i nuclei intensamente colorati di altrettante difefrenti cellule. In ciascuna cellula è contenuta una molecola di DNA, identica se le cellule stesse provengono dallo stesso individuo. Si tratta di estrarre l’informazione da quei piccoli scrigni biologici che contengono l’informazione della vita, il DNA.
7.2.1 L’estrazione del DNA
Art. 19 (reg. att. Banca dati) – Per l’eventuale fase di estrazione del DNA dai campioni biologici sono utilizzati kit commerciali di uso comune nell’ambito della comunità forense e validati dalla letteratura scientifica in termini di resa quantitativa e qualitativa del DNA estratto.
Trasferito in una provetta assolutamente DNAfree, il materiale è sottoposto a una procedura che permette di liberare il DNA dal nucleo e dalle membrane. Contemporaneamente deve essere creato un ambiente nel quale il materiale genetico sia conservato in condizioni ottimali, per garantirne la stabilità. Una buona preparazione di DNA è alla base dell’efficacia di qualunque analisi di biologia molecolare. Da notare che durante la fase di estrazione vengono irrimediabilmente distrutte tutte le membrane, anche quelle dei mitocondri. Le preparazioni finali contengono dunque, miscelati tra loro, tutti i tipi di DNA. Sia nucleare che mitocondriale, sia tutti i tipi di RNA presenti nella cellula. Si utilizzano svariate tecniche a seconda del tipo di matrice. Tutte includono soluzioni tamponate, con additivi quali detergenti, stabilizzanti e una sostanza particolare, detta proteinasi K. Questa ha la funzione di digerire le proteine e inattivare le nucleasi capaci di degradare l’esile filamento del DNA.
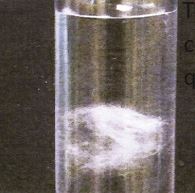
L’esito della fase di estrazione di materiale genetico porta a una soluzione incolore nella quale il DNA è completamente solubilizzato. In alcuni casi si passa alla purificazione degli estratti e alla loro eventuale concentrazione, così da raccogliere il DNA in un piccolo volume.
Tuttavia in condizioni particolari e in presenza di grossi quantitativi il DNA può essere precipitato. Considerando che è insolubile in alcool, formerà una piccola massa gelatinosa di aspetto diafano che quindi può essere visualizzata.
Esistono procedure diverse di estrazione a seconda delle matrici. Anche per questo è utile avere informazioni circa la natura della traccia, prima di procedere a queste fasi. Per esempio, reperti ossei dovranno essere pretrattati con una sostanza in grado di rimuovere i depositi di calcio. Solo così si potrà accedere agli osteociti, le cellule del tessuto osseo, all’interno delle quali sono contenute le molecole di interesse.
La digestione di cellule fortemente cheratinizzate, come unghie e capelli, richiede poi l’impiego di sostanze additive. Tra queste il ditiotreitolo (DTT), in grado di distruggere i ponti disolfuro delle proteine dei peli e delle membrane degli spermatozoi. Sfruttando queste differenze nella sensibilità a certe sostanze chimiche è anche possibile impiegare metodiche differenziali di estrazione. Così si possono talvolta separare componenti cellulari, con caratteristiche diverse, siano mescolate insieme.
L’analisi individuale di tracce miste rappresenta infatti una difficoltà ed è sempre preferibile, se possibile, ridurne la complessità, isolando le diverse componenti cellulari. Il tipico esempio è costituito dai reperti da violenze sessuali. In questi casi si tratta prevalentemente di tamponi assunti alla vittima durante la visita in ospedale, contenenti sia spermatozoi dell’aggressore che, naturalmente, cellule dell’epitelio femminile. Usando una metodica di estrazione differenziale è possibile isolare la sola componente femminile, e solo successivamente estrarre quella maschile, disgregando le membrane degli spermatozoi.
Per le fasi successive, il regolamento indica la possibilità dell’impiego di metodologie che aiutino l’analista nella gestione dei campioni. All’art. 20, «Preparazione dei campioni con sistemi robotizzati», infatti, si dà facoltà dell’utilizzo di metodiche automatizzate per le fasi di preparazione del campione (quantificazione, amplificazione e caricamento su sequenziatore automatico), al fine di ridurre l’errore umano. Il sistema automatizzato deve avere idonea documentazione IQ/OQ o equivalente o superiore che ne dimostri la corretta installazione e funzionamento.
3.2. La quantificazione del DNA
Art. 21 (reg. att. Banca dati) – Per l’eventuale fase di quantificazione del DNA sono utilizzati kit commerciali che consentono di verificare la quantità del DNA presente nell’estratto e la presenza di eventuali inibitori della PCR.
Sui campioni ordinari non troppo esigui, come macchie di saliva, tracce di sangue, tessuti, la quantificazione può essere effettuata con metodi semplici. Questi sfruttano la capacità della molecola del DNA di assorbire la radiazione elettromagnetica di preferenza nell’ultravioletto, a 260 nm. Quando si abbia a che fare con tracce ridotte, comuni per i reperti forensi, si debbono utilizzare tecniche più sofisticate. È oggi molto diffusa la PCR quantitativa, metodo analitico che consente di dosare, teoricamente, fino a una singola cellula di DNA umano.
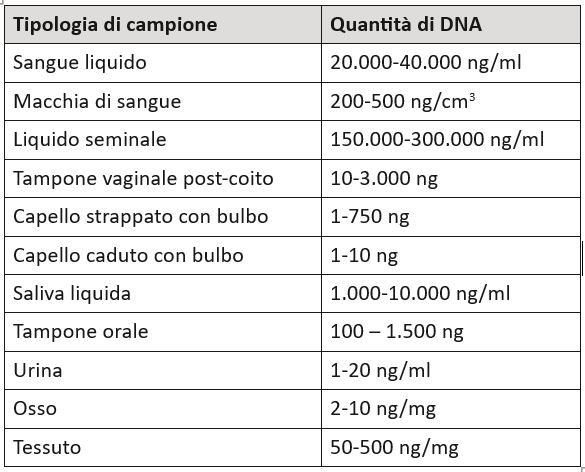
In linea di massima la quantità di DNA è direttamente proporzionale al numero di cellule contenute in una traccia. Ma dipende molto dalla tipologia del materiale biologico, come mostra la tabella a lato.
La verifica quantitativa è attività imprescindibile nel processo di tipizzazione. E’ una necessità contenuta in una raccomandazione dell’ISFG del 1992 [2], e questo sostanzialmente allo scopo di effettuare analisi più precise.
In condizioni ideali il materiale da analizzare dovrebbe essere abbondante e purificato. Nella pratica vista la natura dei vari reperti è più probabile avere a che fare con matrici scarse e inquinate. Tra gli inibitori, ci sono sostanze naturali proteiche, come l’ematina e la cheratina, e sintetiche costituite dai vari substrati con i quali il campione può essere miscelato, asfalto, gesso, coloranti, ecc. Gli esami con il metodo quantitativo indicano con precisione se queste sostanze potranno ostacolare il processo analitico successivo. Indirizza pertanto l’analista a utilizzare tutte le tecniche disponibili per la loro rimozione.
3.3. L’amplificazione del DNA
Art. 22 (reg. att. Banca dati) – Amplificazione del DNA.
1. Lo strumento impiegato è un thermal cycler con certificazione sull’affidabilità dei cicli delle temperature utilizzate dal kit di amplificazione del DNA.
[…]
- L’amplificazione di ogni singolo campione biologico deve essere effettuata attraverso l’uso di due kit commerciali che hanno per il medesimo locus una diversa sequenza dei primers, al fine di evitare una non corretta assegnazione allelica.
- I loci amplificati dai due kit commerciali si devono sovrapporre per almeno dieci loci.
- L’amplificazione del DNA deve essere allestita con il controllo positivo presente nel kit ed un controllo negativo.
Il fine ultimo dell’analisi del DNA è dunque quello di accertare quale sia il profilo genetico del campione/reperto. Si descrive esattamente, per ciascun marcatore, quali siano gli alleli che lo compongono. Con la fase di estrazione il DNA è stato liberato dalle cellule e purificato. La fase di quantificazione è stata necessaria per capire quanto ne è stato ottenuto. In ogni caso, è veramente troppo poco per poterlo studiare con metodi ordinari.
È qui che entra in gioco la vera innovazione tecnologica del test genetico. Un sistema artificiale di tipo biochimico, dunque in vitro, attraverso il quale è possibile generare la replicazione esponenziale dei frammenti del DNA contenenti i marcatori da analizzare. Il tutto in una provetta di plastica di un paio di centimetri.
Come ricorda anche la norma, si parla di «amplificazione del DNA». La tecnica che vi sottende è conosciuta con il nome di «Reazione a catena della polimerasi» (Polimerase Chain Reaction o PCR). Le applicazioni di questo metodo sono numerose in tutti i settori delle scienze della vita, dalla medicina, alla paleontologia, alla biologia, alla genetica.
Fu ideata da Kary Mullis, al quale per questo fu conferito il premio Nobel per la chimica nel 1993[3]. Il funzionamento teorico della PCR è straordinariamente semplice e, proprio per questo, geniale. Mullis lavorava alla Cetus, una compagnia americana, a un progetto per l’analisi di una mutazione genetica che provoca l’anemia falciforme. C’era il problema che lavorare su poco DNA produceva un grave ritardo nel fornire risposte ai genitori dei bimbi interessati alla diagnosi. Egli scoprì che era possibile riprodurre artificialmente molte copie del materiale genetico da studiare. Qualcosa di simile a ciò che le cellule fanno abitualmente all’interno dell’organismo, la duplicazione del proprio DNA. Il tutto utilizzando un enzima, una polimerasi artificiale, estratto da un batterio, e ricreando in una provetta un ambiente simile al nucleo cellulare.
Selezionando primer specifici, cioè delle brevi sequenze di DNA, è in effetti possibile indirizzare l’azione di una polimerasi artificiale in corrispondenza della zona di interesse. SI va letteralmente a «pescare», all’interno dei tre miliardi di paia di basi di cui si compone il DNA. Un frammento di poche centinaia di basi che rappresenta l’oggetto dello studio, contenendo il marcatore.
Ciò che è straordinario è che si tratta di una «pesca automatica», nel senso che una volta predisposta la reazione, essa avviene senza alcun intervento dell’analista. Procede per mezzo di una serie consequenziale di cicli termici in ognuno dei quali il prodotto del ciclo precedente funge da stampo per quello successivo. Trattandosi di un metodo basato su cicli esponenziali, sono sufficienti poche copie iniziali originate da poche cellule, per generare miliardi di copie del frammento contenente il marcatore che si è scelto di analizzare.
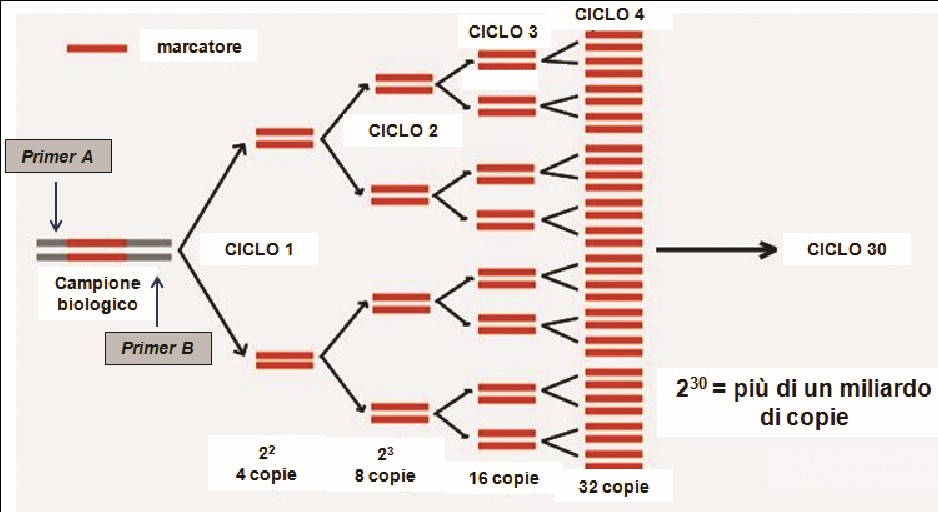
Quando sono disponibili basi teoriche così robuste, la tecnologia è in grado di aiutare in concreto gli analisti nella conduzione delle proprie attività. Così, i primi esperimenti di Mullis prevedevano per ciascun esperimento che la provetta fosse trasferita manualmente, per ogni ciclo, in bagnomaria. Usando temperature diverse, ripeteva i passaggi al ciclo successivo, e così via. Tutto questo oggi si fa con piccole macchine dedicate e certificate riguardo al perfetto funzionamento. Sono chiamate termociclatori (thermal cycler), molto rapide, tanto che un intero ciclo di PCR si effettua in circa un’ora e mezzo.
Esiste un’ampia gamma di termociclatori con differenti performances che rendono relativamente semplice l’esecuzione della PCR. È fondamentale che vi sia uniformità tra le temperature impostate e quelle effettivamente raggiunte. La loro taratura e controllo periodico è necessaria. L’amplificazione può essere effettuata in provette singole oppure in piastre, per l’analisi contemporanea di molti campioni alla volta.
Non è indispensabile poi analizzare un marcatore alla volta, anzi la disponibilità di kit commerciali consente oggi di studiarne molti contemporaneamente, con le reazioni dette multiplex. Queste sono concepite in maniera tale da contenere cocktail di primer per le diverse regioni polimorfiche. L’impiego di una reazione multiplex soddisfa diverse necessità:
- è sufficiente una quantità ridotta di DNA per avere informazioni da molti marcatori genetici contemporaneamente, ottenendo quindi un’elevata informatività in una sola reazione;
- possono essere studiati insieme tutti i marcatori del CODIS o dell’ESS per lo scambio dei risultati delle analisi del DNA;
- è facile individuare immediatamente la presenza di misture in base al numero di alleli osservati per locus.
Esistono diversi kit distribuiti da ditte specializzate concepiti in modo simile, con differenze riguardo alla chimica della reazione e alle sequenze dei primer utilizzati. Quest’ultimo aspetto è sottolineato dal regolamento che impone, al fine della definizione del profilo genetico, che ciascun campione biologico sia analizzato con due kit commerciali diversi, che abbiano tuttavia almeno dieci marcatori (loci) in comune.
Altro aspetto richiamato dalla normativa è quello dell’uso di controlli interni. Quando viene effettuata l’analisi di un campione, è sempre necessario esaminare nella stessa sessione almeno due tipologie di controlli:
- un controllo negativo che contenga tutti gli ingredienti della reazione, ma non DNA (l’esito dell’esame non deve produrre alcun profilo, ancorché parziale).
- un controllo positivo che contenga un DNA noto di cui, a priori, sia noto il profilo genetico (l’esito dell’esame deve confermare l’assetto atteso);
L’uso di questi controlli, specialmente di quelli negativi, è essenziale, con molte implicazioni pratiche per il controllo globale del processo.
La fase dell’amplificazione produce dei quantitativi enormi di DNA. Per questo motivo che le zone del laboratorio dove si effettua la PCR e dove sono mantenute le provette con le reazioni, devono essere assolutamente separate dalle altre zone del laboratorio, con accessi controllati che impediscano le contaminazioni.
È evidente dunque che l’analista effettivamente non esamina il DNA originale della traccia, ma una sua riproduzione artificiale. Fondamentale è allora la capacità dell’intero metodo, e in particolare del sistema di amplificazione, di generare copie affidabili del DNA originale. Centinaia di migliaia di esperimenti condotti in laboratori di tutto il mondo e i continui processi di validazione, dimostrano che il metodo funziona. Gran parte del successo della genetica forense deriva dalla sua robustezza.
L’esito della fase di amplificazione produce quindi una quantità enorme di frammenti di DNA, ma ancora assolutamente invisibili e dispersi in una matrice incolore. Per vedere cosa sia contenuto in quella provetta occorre un altro sistema analitico: l’elettroforesi.
3.4. L’elettroforesi del DNA
Art. 23 (reg. att. Banca dati) – Lettura ed interpretazione del profilo di DNA.
- La determinazione del profilo genetico deve avvenire utilizzando un sequenziatore automatico di acidi nucleici per la corsa elettroforetica dei frammenti di DNA e dotato di software dedicati alla successiva lettura ed interpretazione dei profili del DNA.
- Per una corretta assegnazione allelica devono essere seguiti i seguenti criteri minimi standard;
- ogni corsa elettroforetica deve avere almeno un ladder allelico;
- in ogni corsa elettroforetica a ciascun campione di PCR deve essere associato uno standard interno di peso molecolare noto;
- l’assegnazione degli alleli di un profilo del DNA deve essere fatta solo se tutti i picchi dello standard di cui alla lettera b) sono stati correttamente assegnati;
- l’assegnazione degli alleli di ciascun profilo del DNA viene effettuata mediante software specifici;
- l’interpretazione del profilo del DNA deve essere effettuata da due persone distinte o dalla stessa persona in momenti diversi;
- i picchi denominati «stutter» devono avere un’altezza media non superiore al valore percentuale stabilito dalla validazione del kit utilizzato per quel locus;
- tutti i picchi dell’elettroferogramma devono essere distinti dalla linea di base.
Il risultato dell’amplificazione dei marcatori è la produzione di frammenti che differiscono, gli uni dagli altri, in base alla lunghezza. Si utilizzano in laboratorio degli strumenti detti «sequenziatori automatici di acidi nucleici». Sono in grado di determinare con estrema precisione la dimensione dei frammenti generati a seguito della reazione a catena della polimerasi. I sequenziatori ormai universalmente utilizzati in genetica forense sono del tipo capillare. Garantiscono analisi rapide anche di molti campioni in una singola sessione (detta «corsa» o «sessione elettroforetica»).
Le varie forme alleliche sono individuate perché i primer utilizzati per condurre la reazione di PCR sono marcati, all’origine, con dei fluorocromi fluorescenti. Hanno massimi di emissione a determinate lunghezze d’onda, quando eccitati da una sorgente laser. Il rivelatore del sistema elettroforetico, un apposito detector, è in grado di leggere i segnali emessi, trasformandoli mediante il software di analisi in picchi elettroforetici. Esistono vari modelli di sequenziatori, con numero variabile di capillari, sottilissimi tubicini dello spessore di pochi micron all’interno dei quali avvengono le separazioni degli amplificati. I moderni strumenti sono oggi in grado di leggere fino a 8 fluorocromi, il che permette di identificare un gran numero di marcatori del DNA.

I dati grezzi (detti raw data) ottenuti da questi sistemi di rilevazione sono costituiti da una serie di picchi dai colori virtuali che ne consentono l’interpretazione, mediante un idoneo software associato allo strumento.
Un tipico tracciato elettroforetico riporta in ascisse le dimensioni dei frammenti di DNA e in ordinate l’intensità di fluorescenza relativa (RFU).
Esiste una soglia di sensibilità che l’operatore deve scegliere, in base a un’apposita procedura di validazione interna, per l’identificazione di ciascun picco allelico. I picchi che superano la soglia verranno riconosciuti dal software di analisi e identificati come alleli; quelli al di sotto potrebbero essere confusi con il rumore di fondo strumentale e non saranno quindi riconosciuti come contributo del genotipo di quel campione.
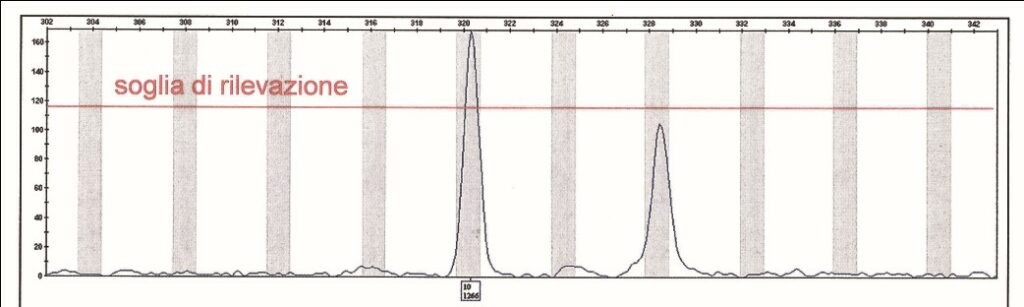
Esempio di profilo allelico a un marcatore. Solo l’allele che supera la soglia (posta in questo caso a 110 RFU) è rilevato dal sistema analitico. L’altro picco, posto al di sotto della soglia analitica non è rilevato nel genotipo di questo campione.
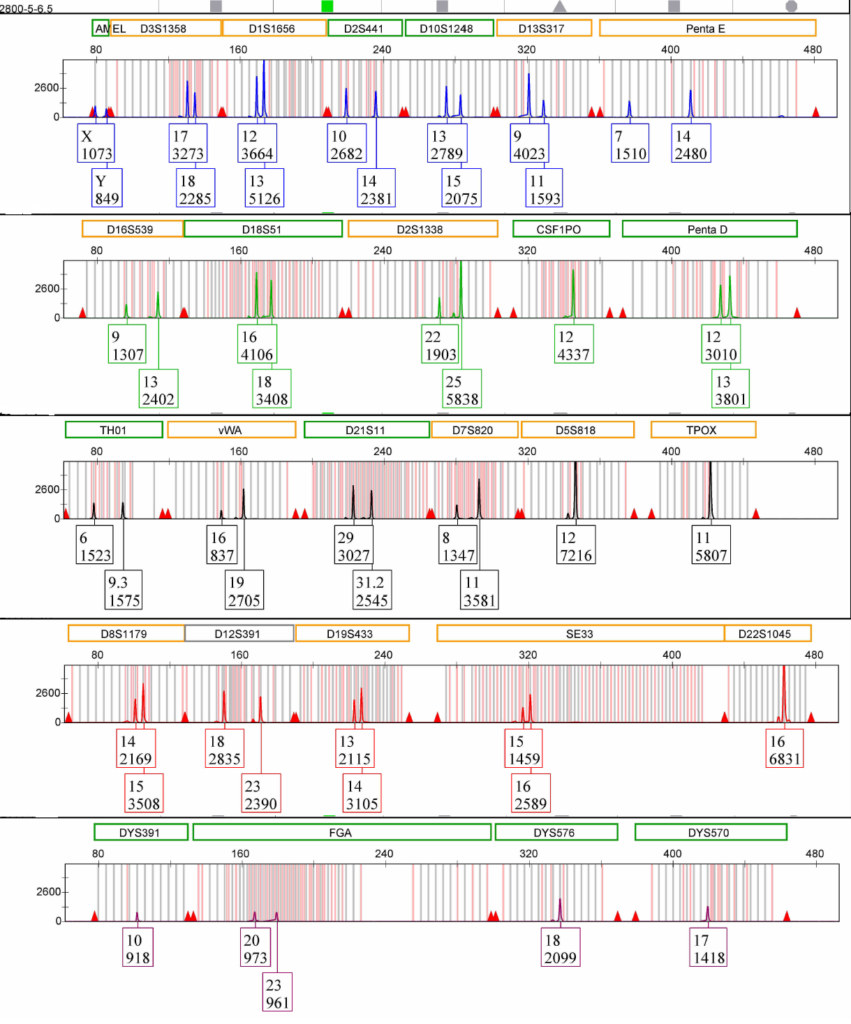
Sulla sinistra un profilo genetico ottenuto da un campione o reperto. Ogni elettroferogramma reca una serie di informazioni che saranno oggetto delle valutazioni dell’analista. Mostra l’assetto di ciascun marcatore con gli alleli già numerati dal software in base alla classificazione internazionale.
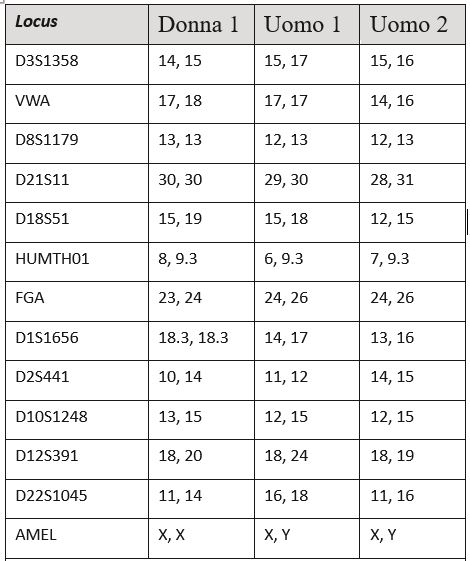
La rappresentazione grafica del profilo genetico è difficilmente maneggiabile, nel senso che si tratta di un dato difficile da scambiare con un altro utente. Si utilizza allora una trasformazione del dato grafico in uno numerico utilizzando delle tabelle. Ciascun locus del DNA è rappresentato da dei numeriche, nel loro insieme, rappresentano la caratterizzazione individuale del campione/reperto.
[1] Questi sono alcuni link: http://www.funsci.com/fun3_it/dna/dna.htm; video su estrazione da banana: https://www.youtube.com/watch?v=2JIgMUXzuy8; video su estrazione da kiwi: https://www.youtube.com/watch?v=_q9SgAEx1Gs.
[2] W. Bär et al., Editorial: Recommendations of the DNA Commission of the International Society for Forensic Haemogenetics relating to the use of PCR-based polymorphisms, in Forensic Sci Int (2002)55, 1-3.
[3] Cf. R. Saiki et al., Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia, in Science (1985)233, 1076-1078; K.B. Mullis – F.A. Faloona, Specific synthesis of DNA in vitro via polymerase-catalyzed chain reaction, in Methods enzymol (1987)155, 335-350.
