Il futuro è molto aperto, e dipende da noi, da noi tutti.
Karl Popper
Dipende da ciò che voi e io e molti altri uomini fanno e faranno, oggi, domani e dopodomani. E quello che noi facciamo e faremo dipende a sua volta dal nostro pensiero e dai nostri desideri, dalle nostre speranze e dai nostri timori. Dipende da come vediamo il mondo e da come valutiamo le possibilità del futuro che sono aperte.
Sono diversi gli aspetti rilevanti che riguardano gli sviluppi della materia. Il primo riguarda la tecnologia, con innovazioni indubbiamente promettenti in termini di capacità analitica. Vi è anche molto spazio per la ricerca, in genetica forense, con opportunità veramente interessanti che potrebbero fornire informazioni particolarmente utili per la ricostruzione storica di un fatto. Nel momento in cui scrivo queste righe l’attenzione di tutti è però diretta alla tematica dell’accreditamento dei laboratori, una necessità oggi cogente per poter alimentare la Banca dati del DNA e soprattutto per lavorare a un certo livello in questa disciplina. In ogni caso, sia che si tratti di ricerca che di attività analitica, si tratta di opportunità di miglioramento per lavorare al meglio.
Gli investigatori hanno interesse ad acquisire più informazioni possibili da una traccia, per risalire a chi l’abbia lasciata in modo da dare un volto al donatore sconosciuto con il fine dichiarato di risalire alla sua identità.

Qualche anno fa alcuni articoli destarono un certo scalpore dichiarando come fosse possibile risalire da un reperto biologico al «cognome» di chi avesse lasciato il campione sulla scena del crimine. Sì, avete letto bene. Il fatto è che il cognome, nelle società occidentali, si trasmette con le stesse modalità del cromosoma Y, cioè attraverso una trasmissione unidirezionale paterna. Così, se fosse stato possibile associare a ciascun aplotipo Y il cognome del relativo possessore, sarebbe anche stato possibile individuare, facendo il percorso al contrario, da un aplotipo Y il cognome del donatore di una traccia [1].
Anche nel laboratorio dove lavoravo a quei tempi provammo con entusiasmo a esaminare un campione di 107 uomini [2], con il risultato di trovare effettivamente una notevole diversità per 106 aplotipi, risultati differenti, confermando dunque l’informatività degli Y-STR, e individuando due identici genotipi di persone non imparentate, ma che risultarono avere lo stesso cognome. Tuttavia, nella stessa popolazione che esaminammo, vi erano tre coppie di uomini non correlati, con lo stesso cognome, ma con differenti aplotipi del cromosoma Y a sottolineare che questo assioma diretto ha uno scarso fondamento reale. Si tratta effettivamente di un metodo che non trova applicazioni pratiche, anche se in genetica forense è bene aggiungere sempre la parola «finora».
La ricerca delle caratteristiche somatiche dal DNA è un altro aspetto sul quale sono stati sviluppati, negli anni, molti studi che hanno però condotto a individuare con una certa sicurezza pochi tratti caratterizzanti gli individui. Il fenotipo è dovuto infatti non al prodotto di un singolo gene, ma a quello di molte proteine ed enzimi ed è la loro reciproca interazione che determina lo sviluppo dei caratteri somatici. Si tratta quindi di «caratteri multifattoriali» e le corrispondenze genotipo-fenotipo si riscontrano solo in casi particolari.
Studiando alcuni tratti del recettore della melanocortina (MC1R) si è visto che è possibile predire con elevata accuratezza se una persona abbia i capelli di colore rosso [3]. Altri studi hanno poi dimostrato che è possibile stabilire il colore degli occhi, esaminando alcune varianti alleliche su un certo numero di geni [4], con la limitazione che la variabilità fenotipica è talmente elevata nelle numerose sfumature delle iridi, da renderle difficilmente classificabili. Un’altra possibilità è quella di accertare il colo re della pelle, anche in questo caso analizzando delle variazioni a singolo nucleotide (SNP), in combinazione con l’esame per il colore degli occhi e dei capelli [5].
Un’altra informazione importante per le investigazioni riguarda l’origine etnica del donatore di una traccia. Questo tipo di analisi presenta un limite. Le persone tendono ormai a un elevato inbreeding, con un aumento dei matrimoni misti e dunque con la conseguenza che il concetto stesso di popolazione ha contorni sfumati [6]. Tuttavia, studiando una serie di polimorfismi, è possibile avere indicazioni sulle origini geografiche di chi ha lasciato una traccia. Si tratta però di inferenze di massima che consentono di differenziare macropopolazioni (Eurasia, Africa subsahariana, Sud-Est asiatico, ecc.), ma con scarsa risoluzione per le popolazioni continentali.
Ora, tutte queste applicazioni sono disponibili in un moderno laboratorio di genetica forense. Ma il loro impiego è limitato a casi particolarmente complessi, non rientrando certamente nella routine quotidiana. Si tratta di applicazioni che seguono i primi esami volti alla determinazione del profilo genetico, irrinunciabili, quelli sì, per l’identificazione.
Nel frattempo le nuove tecnologie, in particolare quelle di sequenziamento massivo, stanno facendo capolino nel settore forense. Si tratta degli strumenti di Next-Generation Sequencing (NGS). Introdotti nella metà dell’anno 2000 e in grado di sequenziare contemporaneamente migliaia di campioni biologici in centinaia di geni. Con queste piattaforme è possibile acquisire una quantità straordinaria di informazioni da un campione biologico, in tempi contenuti. Quello che i ricercatori hanno impiegato decine di anni per portare a compimento, grazie al lavoro di migliaia di ricercatori in tutto il mondo, e cioè il sequenziamento dell’intero genoma umano, oggi può essere fatto da un singolo operatore nel giro di pochi giorni, con costi contenuti.
Si assiste quindi quasi a un paradosso. Si producono grandi quantità di dati strumentali al punto che è stato necessario affiancare, alla classica figura dell’analista di laboratorio, quella dell’analista informatico. In modo da poter interpretare l’enormità dei dati emergenti. In genetica medica questo eccesso d’informazioni ha destato alcune perplessità. Effettuando esami diagnostici contemporaneamente su un numero elevato di geni, si ottengono risultati anche su quelli per i quali non fosse stato proposto un quesito diagnostico. Traslando questo tipo di problematiche verrebbe quindi da chiedersi come gestire dati di questa natura. Quando acquisiti su un reperto forense da cui ci si attende di avere solo informazioni per l’identificazione personale.
Le principali società commerciali del settore stanno investendo nel settore. Sono già disponibili sistemi standardizzati per analisi forensi che limitano i dati, estendendoli a quelli con valenza solo identificativa ed escludendo tutti gli altri con valenza medica, genetica e predittiva riguardo a patologie ereditarie. Per esempio, è possibile acquisire contemporaneamente informazioni da un ampio pannello di SNP, utilizzando la piattaforma HID-Ion AmpliSeq™ Identity Panel (Thermo Fisher Scientific). Anche effettuare l’intero sequenziamento dell ’mtDNA, oltre allo studio di marcatori diversi.
Con altri kit è possibile esaminare contemporaneamente una varietà di zone polimorfiche, compresi microsatelliti autosomici, X-STR, Y-STR e un numero elevato di SNP. L’evoluzione è certamente continua e anche compagnie che non producono strumentazioni NGS stanno introducendo sul mercato kit che consentono analisi contemporanee di molte zone polimorfiche.
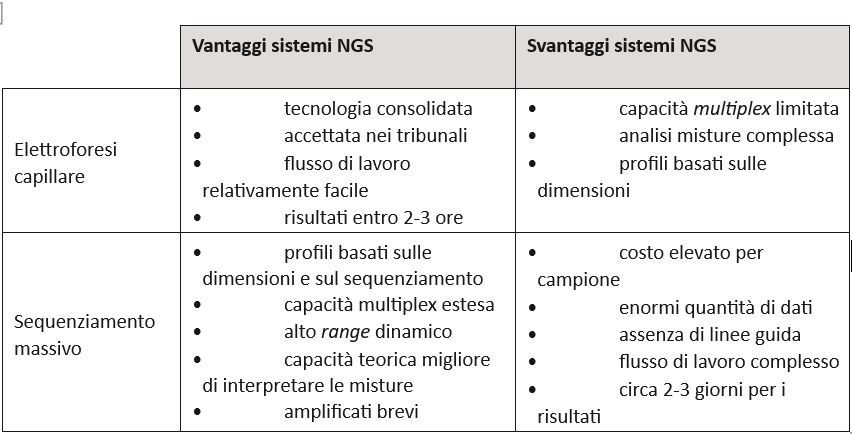
L’idea di poter fornire un «identikit genetico» riguardo alle sembianze di una persona mi pare ancora fantasiosa. Il fatto di poter interrogare simultaneamente molte zone variabili del DNA può realmente essere d’aiuto all’investigatore. Specialmente laddove nessun’altra informazione sia disponibile su chi ha lasciato traccia di sé sul luogo del reato[7]. Tuttavia, è difficile pensare a un uso su larga scala di queste tecnologie, in Italia. La Banca Dati prevede l’impiego di sistemi di elettroforesi capillare e nonè prevista la sostituzione di questa tecnica con i nuovi metodi.
Mentre ci stiamo interrogando su quale delle tecnologie si affermerà nell’immediato futuro, i ricercatori vanno avanti e le lampadine delle idee si accendono. Per esempio, l’idea di poter discriminare tra loro due gemelli identici, impossibile fino a pochi anni fa, potrebbe diventare una realtà. Si tratta di ricercare, proprio come «un ago nel pagliaio» quelle rare differenze riscontrabili nei DNA di gemelli monozigoti [8]. Dovute a rarissime mutazioni somatiche prodotte nelle prime duplicazioni postzigotiche possono servire a stabilire differenze tra fratelli/sorelle. E’ noto che ci sono più dell’83% di probabilità che un nascituro di una coppia di gemelli monozigoti sia portatore di una mutazione. Questa differenza genetica rispetto al proprio fratello gemello può essere usata per l’individualizzazione [9]. Ne ho parlato anche nell’ultimo libro che ho scritto i cui riferimenti troverete in questo sito.
I contributi delle nuove tecnologie si estendono poi a una straordinaria e vasta gamma di settori addizionali. Tra questi la possibilità di identificare le cause di morte improvvisa e la risposta inappropriata a farmaci. Si apre dunque al settore dell’«autopsia molecolare». Qui la biologia molecolare può davvero fornire un contributo sostanziale per la comprensione di eventi altrimenti inspiegabili [10].
Nonostante la genetica forense sia prevalentemente una scienza applicativa, vi sono molte aree di interesse che necessitano di approfondimento e per le quali il contributo della ricerca è essenziale. Basti pensare all’importanza dell’esatta identificazione dei fluidi biologici, propedeutica ai test del DNA e alla diagnosi temporale, sia riguardo all’epoca di deposizione di una traccia che a quella, forse più importante, dell’epoca della morte.
[1] B. Sykes – C. Irven, Surnames and the Y Chromosome, in Am J Hum Genet (2000)66, 1417-1419.
[2] U. Ricci et al., Y-chromosomal STR haplotype in Tuscany (central Italy), in Forensic Sci Int (2001)120, 210-212.
[3] W. Branicki et al., Model-based prediction of human hair color using DNA variants, in Hum Genet (2011)129, 443-454.
[4] S. Walsh et al., Developmental validation of the IrisPlex system: determination of blue and brown iris colour for forensic intelligence, in Forensic Sci Int: Genet (2011)5, 464-471.
[5] P. Dario, Assessment of IrisPlex-based multiplex for eye and skin color prediction with application to a Portuguese population, in Int J Legal Med (2015)129, 1191-1200.
[6] J. Jia et al., Developing a novel panel of genome-wide ancestry informative markers for bio-geographical ancestry estimates, in Forensic Sci Int: Genet (2014)8, 187-194.
[7] M. Kayser – P. de Knijff, Improving human forensics through advances in genetics, genomics and molecular biology, in Nat Rev Genet (2011)12, 179-192.
[8] J. Weber-Lehmann et al., Finding the needle in the haystack: differentiating “identical” twins in paternity testing and forensics by ultra-deep next generation sequencing, in Forensic Sci Int: Genet (2014)9, 42-46.
[9] M. Krawczak et al., How to distinguish genetically between an alleged father and his monozygotic twin: a thought experiment, in Forensic Sci Int: Genet (2012)6, 129-130.
[10] B. Budowle – A. van Daal, Extracting evidence from forensic DNA analysis: future molecular biology directions, in Biotechniques (2009)46, 342-350.
